Développer de nouveaux antibiotiques grâce au microbiote intestinal
Comprendre les mécanismes enzymatiques des bactéries du microbiote intestinal pour développer de nouvelles molécules contre l’antibiorésistance, c’est là l’un des chevaux de bataille d’Olivier Berteau au sein de l’Institut Microbiologie de l’alimentation au service de la santé (MICALIS - Université Paris-Saclay, INRAE, AgroParisTech). Ce spécialiste de la biochimie du microbiote bénéficie pour la troisième fois d’une bourse ERC, dans la catégorie Proof of concept, pour son projet ENTRAP.
La résistance des bactéries aux antibiotiques, aussi appelée antibiorésistance, est devenue une question de santé publique majeure, car responsable de dizaines de milliers de décès par an en Europe. Rien que pour la France, les résistances microbiennes sont responsables d’au moins 5 500 morts par an. Le phénomène de résistance est notamment lié à l’utilisation abusive des antibiotiques. En 2015, l’Organisation mondiale de la Santé (OMS) alerte sur la nécessité d’optimiser l’usage des médicaments antimicrobiens mais aussi de renforcer la recherche et le développement de nouveaux composés efficaces. Un sujet auquel Olivier Berteau et son équipe Chimie et biologie synthétique (ChemSyBio) de l’Institut MICALIS dédient leurs travaux, via l’étude des bactéries du microbiote intestinal.
Un problème de santé au scénario alarmant
L’antibiorésistance correspond au fait qu’un traitement antibiotique ne soit plus efficace contre une infection bactérienne. Classiquement, un traitement antibiotique neutralise les bactéries responsables de l’infection, et seule une quantité mineure de bactéries résistantes survit dans l’intestin. Or l’administration répétée d'antibiotiques ou leur usage inadapté peut mener à la prédominance des bactéries résistantes. La surutilisation des antibiotiques, en particulier, génère une « pression de sélection » dans l’organisme qui favorise les mutations et la reproduction de ces bactéries.
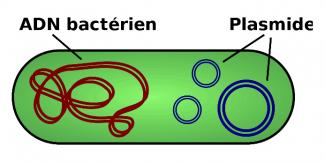
Lorsque le phénomène de résistance vient de mutations chromosomiques sur l’ADN bactérien, la résistance ne s’applique qu’à un seul antibiotique ou une seule famille d’antibiotiques. Mais dans 80 % des cas, l’antibiorésistance est liée à l’acquisition de matériel génétique par « conjugaison », c’est-à-dire par échange de plasmide d’une bactérie donneuse à une bactérie receveuse. L’information génétique portée par le plasmide, comme des gènes antibiorésistants, peut alors se transmettre de bactérie à bactérie.
Source : Normand.cyr
Si la résistance plasmidique concerne plusieurs antibiotiques, voire plusieurs familles d’antibiotiques, les bactéries sont alors dites multirésistantes. Le transfert de résistance par les plasmides peut se faire entre différentes souches et espèces de bactéries. L’accumulation de mécanismes de résistance chez une même souche bactérienne génère alors une toto-résistance, c’est-à-dire une résistance à l’ensemble des antibiotiques disponibles, conduisant les médecins dans une impasse thérapeutique.
Décrypter le microbiote intestinal
Le microbiote intestinal regroupe l'ensemble des micro-organismes qui colonisent l’intestin grêle et le colon de l’être humain, soit 1012 à 1014 bactéries, virus, parasites et champignons non pathogènes ! C’est le vivier de micro-organismes le plus important chez l’être humain et une ressource encore inexploitée de composés bioactifs et d'antibiotiques, essentiels à son équilibre. Ce potentiel n’a pas échappé à la communauté scientifique et de nombreux projets visant à explorer les capacités digestive, métabolique, immunitaire ou encore neurologique du microbiote intestinal ont vu le jour au cours des dernières années.
Un des axes de recherche de l’Institut MICALIS est justement de comprendre le rôle des bactéries qui colonisent le tube digestif, leur interaction avec leur hôte et leur impact sur la physiologie et la santé humaine. Au sein de l’Institut, l’équipe ChemSyBio étudie plus spécifiquement la synthèse naturelle de peptides bioactifs issus d’enzymes de bactéries situées dans le microbiote intestinal humain.
Dès 2013, Olivier Berteau qui coordonne les travaux de l’équipe reçoit un financement de l’Union européenne, une bourse ERC Consolidator, pour approfondir ses recherches autour des stratégies enzymatiques d’une famille de protéines très polyvalentes, appelées « Radical SAM ». Ces métalloenzymes catalysent une grande variété de réactions, complexes et originales. « Depuis cinq à dix ans, leur importance est de plus en plus mise en évidence dans les voies de biosynthèse de produits naturels, dont des RiPPs (Ribosomally Synthesized and Post-translationally modified Peptides) », explique Olivier Berteau. Les RiPPS naturellement présents dans le microbiote présentent une grande diversité structurale et fonctionnelle, dont, pour certains, des propriétés antibiotiques.
Deux bourses ERC Proof of Concept et un peptide aux propriétés antimicrobiennes prometteuses
En 2017, Olivier Berteau reçoit une bourse ERC Proof of concept pour le projet D-Bug qui vise à transformer les découvertes de l’équipe ChemSyBio en application. « Au départ, nous cherchions à comprendre le fonctionnement des enzymes « Radical SAM » et de plus en plus, nous les utilisons pour trouver d’autres RiPPS », raconte le chercheur.
En 2018, les travaux de l’équipe font l’objet d’une valorisation sous la forme d’un dépôt de brevet et en 2020, tandis que le projet D-Bug se poursuit, Olivier Berteau reçoit une seconde ERC Proof of concept pour le projet ENTRAP (Engineering of natural products), qui s’intéresse à une autre famille de RiPPs. « Nous savons que pour répondre à la crise des antibiotiques, plusieurs dizaines de nouveaux composés doivent être développés. En fonction des applications, les antibiotiques n’ont pas les mêmes besoins en termes de propriétés », ajoute Olivier Berteau.
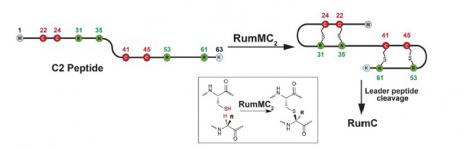
Les travaux les plus récents de l’équipe ChemSyBio portent sur les mécanismes enzymatiques intervenant dans la biosynthèse de la Ruminococcin C (RumC), un RiPP de la catégorie des sactipeptides, produit par l’une des souches bactériennes les plus présentes dans le microbiote intestinal humain, Ruminococcus gnavus. Ce sactipeptide se caractérise par la présence de quatre liaisons α-thioéther intramoléculaires. Introduites par les enzymes Radical SAM putatives, RumMC1 et RumMC2, ces liaisons α-thioéther contraignent les peptides précurseurs C1 ou C2 dans une structure en double épingle à cheveux. Une structure unique, qui associée à l’élimination du peptide signal, confère à RumC une fonction antibiotique active sur un large panel de bactéries, y compris sur des souches multirésistantes aux antibiotiques conventionnels.
Une approche multidisciplinaire pour mieux saisir les mécanismes enzymatiques
Les études biochimiques et mécanistiques de la biosynthèse des RiPP, dont la RumC, sont facilitées par l’arsenal de méthodes utilisées au sein de MICALIS. « Nous combinons différents types d’approches, physico-chimiques, biochimiques, microbiologiques… Nous bénéficions aussi du contexte de l’Université Paris-Saclay pour collaborer avec différents laboratoires », précise Olivier Berteau. Grâce à cette pluridisciplinarité, l’équipe ChemSyBio est capable de caractériser plus précisément les enzymes RumMC1 et RumMC2, ainsi que les intermédiaires réactionnels.
Aujourd’hui, l’équipe continue de déchiffrer ces mécanismes, en tentant notamment de comprendre le rôle des différents groupements identifiés sur les enzymes RumMC1 et RumMC2. La compréhension précise de ces voies de synthèse naturelles est d’autant plus stratégique que RumC possèderait, en plus de sa forte activité antibactérienne, d’autres fonctions d’intérêt thérapeutique comme des propriétés anti-inflammatoires.
Références :
- Clémence Balty, Alain Guillot, Laura Fradale, Clémence Brewee, Benjamin Lefranc, Christian Herrero, Corine Sandström, Jérôme Leprince, Olivier Berteau, Alhosna Benjdia. Biosynthesis of the sactipeptide Ruminococcin C by the human microbiome: Mechanistic insights into thioether bond formation by radical SAM enzymes. J. Biol. Chem. (2020) 295(49) 16665–16677.
- Clémence Balty, Alain Guillot, Laura Fradale, Clémence Brewee, Mylène Boulay, Xavier Kubiak, Alhosna Benjdia, Olivier Berteau. Ruminococcin C, an anti-clostridial sactipeptide produced by a prominent member of the human microbiota Ruminococcus gnavus. J. Biol. Chem. (2019) 294(40) 14512–14525.
- BENJDIA, Alhosna, GUILLOT, Alain, BERTEAU, Olivier. Nouveaux peptides présentant une activité antimicrobienne et nouvelle enzyme apte à convertir un résidu à configuration l en acide aminé à configuration d dans un peptide, Brevet EP3383888 - 10/10/2018.